前言
G蛋白偶联受体(GPCRs)是人类最重要的药物靶点之一,约34%的FDA批准药物通过调控GPCR功能发挥疗效。
然而,GPCR的结构研究长期以来受困于分子量小、构象不稳定等难题,面临巨大挑战。传统方法如T4溶菌酶融合、纳米抗体稳定化等虽有一定效果,但往往依赖大量实验筛选,效率低下。
近日,清华大学刘翔宇团队提出了一种名为 “Click Fusion” 的AI驱动融合蛋白设计策略,实现了对GPCR非激活态的稳定、激活态的锁定,并进一步用于构象特异性纳米抗体的筛选。该策略大部分构建优化工作主要通过计算机实现,减少了对大规模实验筛选的依赖。并且该策略可在短时间内完成从设计到验证的迭代,提高了构建效率。
两项研究成果分别以“De novo design of a fusion protein tool for GPCR research”和“Extracellular nanobody screening using conformationally stable GPCR variants”为题,先后发表于《PNAS》上。


研究内容
1. Click Fusion:非激活态GPCR结构解析的“通用钥匙”
1)非激活态融合蛋白设计流程
团队以目标GPCR的AlphaFold预测结构为模板,利用RFdiffusion从TM5/6 开始,逐步延伸并构建出一个融合蛋白(Clip)骨架。随后,使用ProteinMPNN为这个骨架设计氨基酸序列,再通过AlphaFold2或ESMFold对设计的序列进行结构预测,并以pLDDT分数评估结构合理性和连接区刚性,选择高 pLDDT 分数进入基因合成、蛋白表达与纯化。
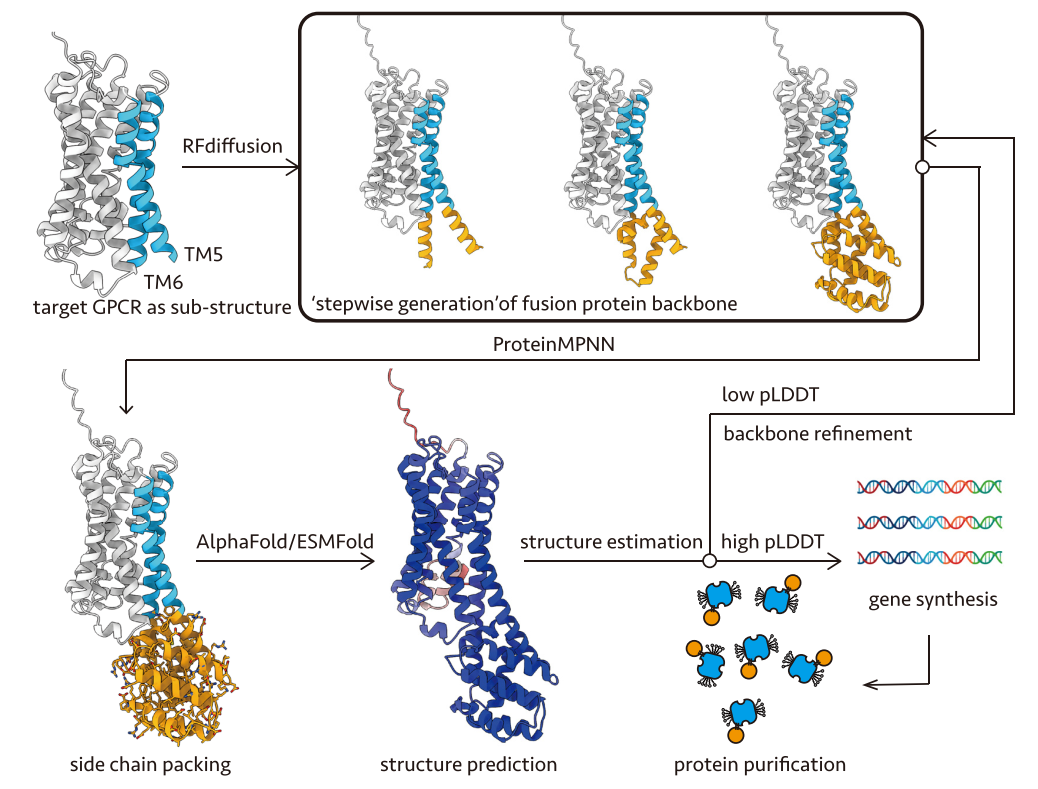
图1. 模式流程图
2)应用成果
成功解析了M1毒蕈碱受体与拮抗剂阿托品复合物(M1R-Clip1)的冷冻电镜结构,分辨率3.3 Å,与AlphaFold预测模型高度一致,且与已知晶体结构没有显著差异。
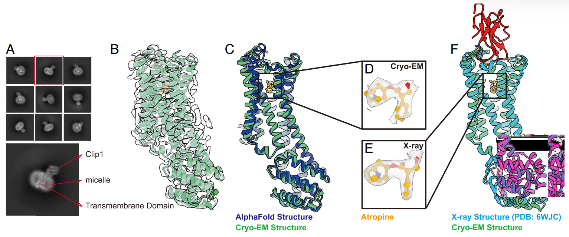
图2. M1R-Clip1的结构分析结果
将该策略应用于长期缺乏非激活态结构的NMBR,成功获得NMBR-Clip2与拮抗剂PD168368的复合物结构,分辨率达3.4 Å。
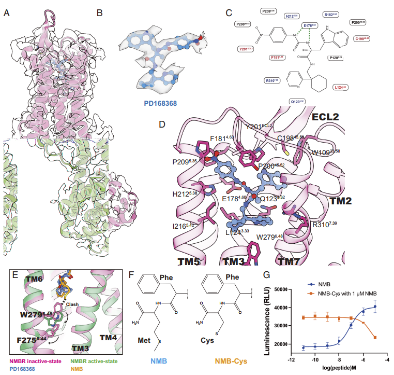
图3. NMBR-Clip的结构分析结果
进一步展示了Clip的“可移植性”:将M1R的Clip1成功迁移至结构相似的5-HT₂BR,首次解析其与拮抗剂balovaptan的复合物结构(3.6 Å),体现了“即插即用”的设计理念。
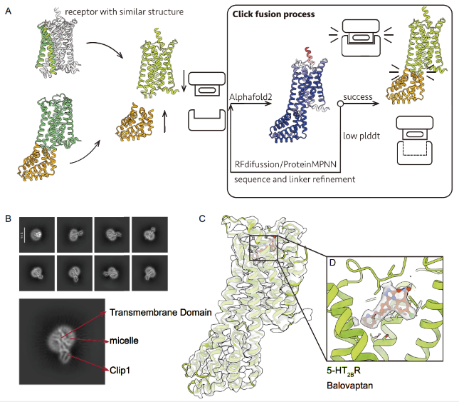
图4. Click Fusion策略和5-HT2BR的整体结构。
2. 激活态锁定+纳米抗体筛选:打开药物开发新维度
1)激活态融合蛋白设计与验证
在第二项研究中,团队进一步拓展融合蛋白设计策略,锁定GPCR于激活态构象。通过将G蛋白α5螺旋(G11CT)与受体C端融合,并在TM5/6间插入设计蛋白,构建出无需激动剂即可维持激活态的M1R-G11CT。
实验表明,该构建体对激动剂iperoxo的亲和力提升约600倍,且冷冻电镜结构显示其在无配体条件下仍处于活性构象,为激活态GPCR的结构与功能研究提供了前所未有的工具。
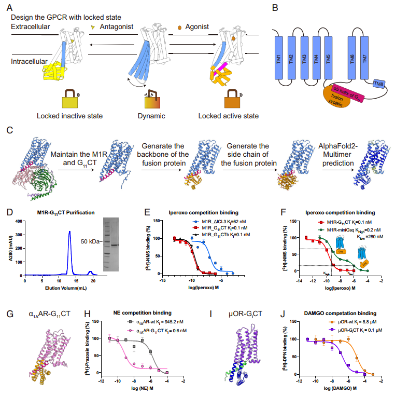
图5. 工程化激活态GPCR构建体的设计与生化表征
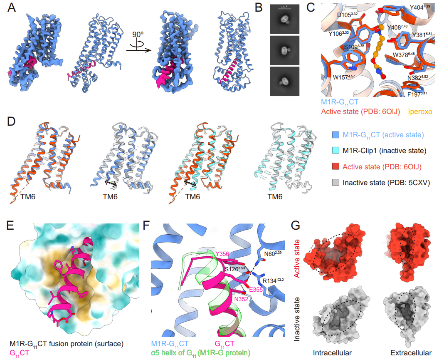
图6. M1R-G11CT的结构揭示了其无激动剂的激活状态
2)构象选择性纳米抗体筛选
基于上述构象锁定平台,团队进行了状态选择性纳米抗体筛选,并提出了“细胞内正交筛选”策略。其核心思想是使用一对或多对“胞外结构完全相同、胞内融合蛋白不同”的受体(如M1R-Clip1与M1R-G11CT)作为诱饵,通过多轮交叉筛选,利用其胞内区的差异来“扣除”结合胞内的抗体,从而定向富集那些识别共同胞外表位的抗体。
通过这一精巧的筛选流程,成功鉴定出了多个具有构象选择性的纳米抗体。并通过冷冻电镜解析其与M1R的复合物结构,揭示其结合于胞外侧变构口袋,具备明确的构象选择性机制。
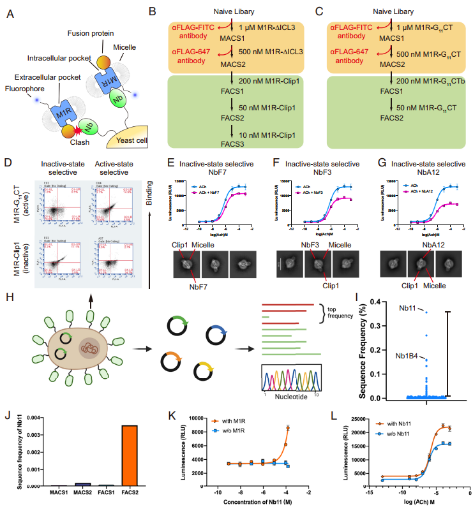
图7. 面向胞外纳米抗体的胞内正交筛选
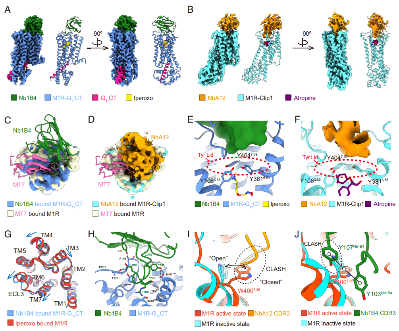
图8. 纳米抗体–M1R复合物的结构
总结
本研究展示了AI在GPCR结构生物学与药物发现中的强大应用潜力。通过“click fusion”策略,我们不仅实现了GPCR非活性与活性状态的高效稳定与结构解析,还开发了一种新型的构象特异性纳米抗体筛选平台。这一方法具有快速、通用、可扩展的特点,为GPCR靶向药物的开发提供了强有力的工具,尤其适用于目前难以成药的GPCR靶点。
未来,该方法有望进一步推广至其他膜蛋白(如转运蛋白),并在结构生物学、抗体工程与药物筛选中发挥更广泛的作用。




















